时空组学技术:生命科学和生物医学的前沿底层工具 | 时空简讯42期
- 来源:华大时空
- 阅读量:1000
- 发表时间:2023-02-27
空间转录组学技术是近年来发展迅速的新兴技术,它能够定量测量细胞的基因表达,追踪细胞在组织空间的精确位置,揭示健康和疾病状态下细胞-细胞相互作用,是生命科学和生物医学领域中重要的技术突破之一。根据如何获取细胞在组织中不同空间位置的信息,该技术主要可分为基于显微切割、原位杂交、原位测序和微阵列技术等类型。为快速、高效地传播、共享有关空间和单细胞多组学技术的开发及应用,华大时空特推出时空/单细胞技术专题简讯。遴选空间和单细胞多组学新技术开发、技术革新与评测类的代表性文献,持续、针对性地进行精要解读,敬请关注。
空间和单细胞多组学技术的发展,能够让我们深入理解健康和疾病状态下细胞的结构与功能。在此,遴选了9篇描述空间转录组技术及其工作原理的优质前沿文章,展示了当前基于不同方法获取细胞空间位置信息的空间转录组学技术进展情况,供了解参考。
综述
Review
空间转录组学技术及其发展方向
Nature Biotechnology [IF:68.164]
① 比较了传统原位杂交定位少量特定分子和空间转录组学(spatial transcriptomics,ST)定量完整组织切片转录组的特点,总结了ST的三个生物学应用场景:阐明组织的细胞类型组成、研究细胞间的相互作用模式和发现不同组织细胞类型间的配体-受体对。
② 将ST技术分为基于测序的ST(sequencing-based ST,sST)和基于成像的ST(imaging-based ST,iST)两大类,并总结了sST和iST的主要应用实例,描述了其表征和验证的关键指标、实验框架,如总结了不同sST技术的空间位置信息的索引生成策略,iST技术原位标记RNA分子的方式和图像处理的主要步骤等。
③ 展望了ST技术未来的发展方向:原位检测转录本剪接、基因组结构变异、表观基因组修饰和调控、蛋白互作和原位蛋白测序,细胞分割和空间分辨率提高下的细胞互作研究,利用基因编辑分子记录技术的四维细胞转录组状态测量。
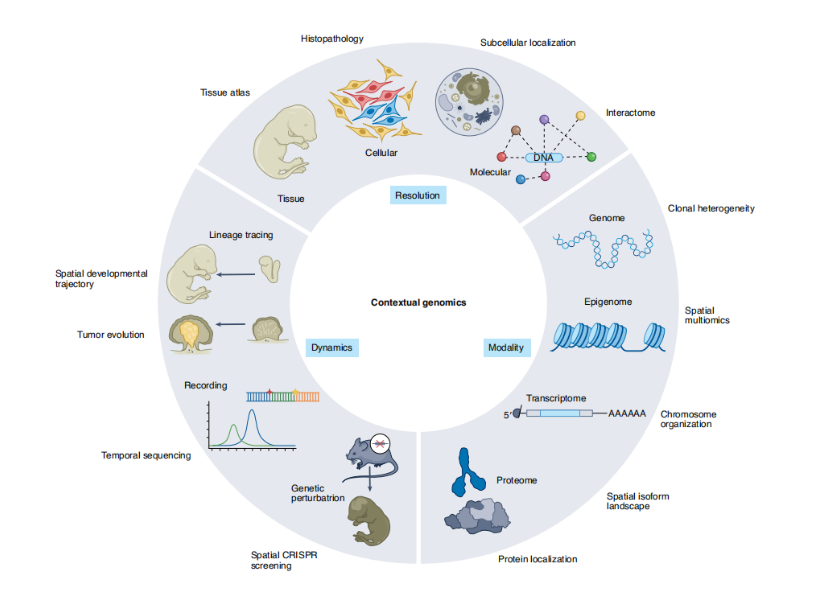
ST技术的发展方向
显微切割技术
Region of Interest Selection
固定组织中RNA和蛋白质的多重数字空间分析
Nature Biotechnology [IF:41.058]
① 描述了数字空间分析(Digital Spatial Profiling,DSP)技术,这是一种适用于福尔马林固定石蜡包埋(FFPE)样品的蛋白质或RNA进行高度多重空间分析的方法,它将传统免疫组化和原位杂交技术发展为一个由硬件、软件和nCounter组成的集成系统。
② DSP依赖于:使用寡核苷酸标签对蛋白质或RNA进行多重读出;通过光裂解(photocleavable,PC)接头连接到亲和试剂(抗体或RNA探针)上的寡核苷酸标签;光切割光投射到组织样本上,在覆盖1~5,000个细胞的感兴趣区域(ROI)中以任何空间模式释放PC寡核苷酸。
③ DSP能够使用抗体读数在ROI内实现单细胞敏感性,RNA检测可适用低至约600个单独的mRNA转录本。
④ 使用nCounter系统显示了淋巴、结直肠肿瘤和自身免疫组织中多达44种蛋白质和96个基因的空间分析,并使用下一代测序展示了1,412个基因的分析。
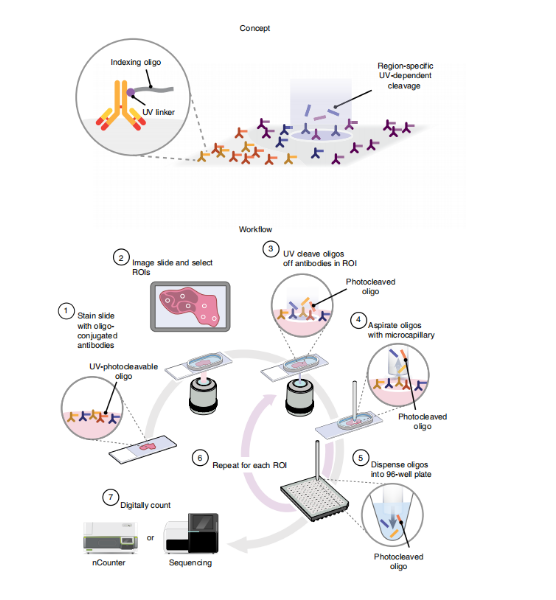
DSP工作原理
Geo-seq:冷冻切片组织样本的空间转录组学分析
Nature Protocols [IF: 12.423]
① 开发了一种结合激光显微切割(LCM)和scRNA-seq的空间转录组技术Geo-seq,允许解析感兴趣区域的少量(10个)细胞的转录组信息,同时还能保留细胞原有位置信息。
② Geo-seq首先对组织进行染色,然后通过LCM感兴趣的细胞,这些切割下来的细胞通过Smart2-seq的方法进行建库并用高通量测序;从组织收集和显微切割到测序的整个流程需要约5 d,数据分析需要1~2周,这取决于数据量和处理器的速度。
③ 使用Geo-seq建立了原肠胚中期小鼠胚胎的3D转录组图谱,并以高精度将单个外胚层细胞映射回体内胚胎位置。此外,Geo-seq还被用于描绘发育过程中小鼠大脑的细胞身份、研究人类精子的病理状态,以及对小鼠肝细胞进行功能表征。
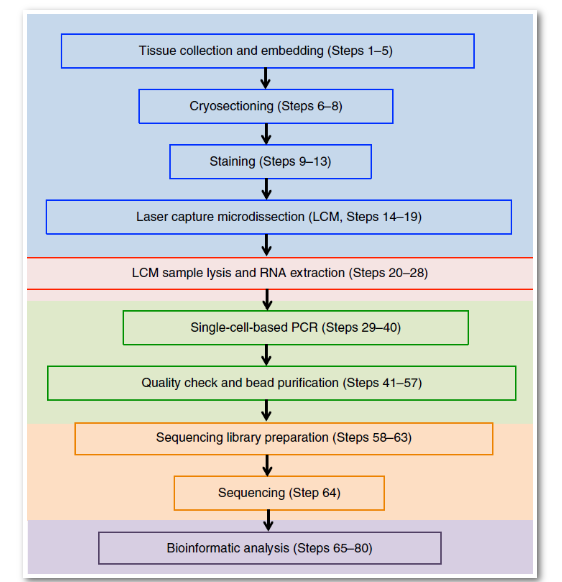
Geo-seq工作流程图
原位杂交技术
Single-Molecule FISH
RNA seqFISH+:实现生物组织中转录组的超分辨成像
Nature [IF:43.070]
① 展示了序贯荧光原位杂交技术的升级版seqFISH+,该技术利用序列杂交和标准共聚焦显微镜成像即可获得单细胞中10,000个基因的超分辨率成像和多路复用。
② 在纯净的NIH/3T3成纤维细胞中检测seqFISH+的实际效率,发现其灵敏度比scRNA-seq更高;进一步使用相同的10,000个基因探针观察小鼠脑室下区皮层和嗅球独立组织切片,总共收集了2,963个细胞中10,000个基因的转录图谱,覆盖面积约为0.5 mm2。
③ seqFISH+能够很好地描述组织中的转录组,克服了光学拥挤,展现了seqFISH+在组织中生成空间单细胞图谱的强大应用潜力;与现有方法相比,seqFISH+可以实现商业共聚焦显微镜的超分辨率成像,并可推广到染色体和蛋白质成像。
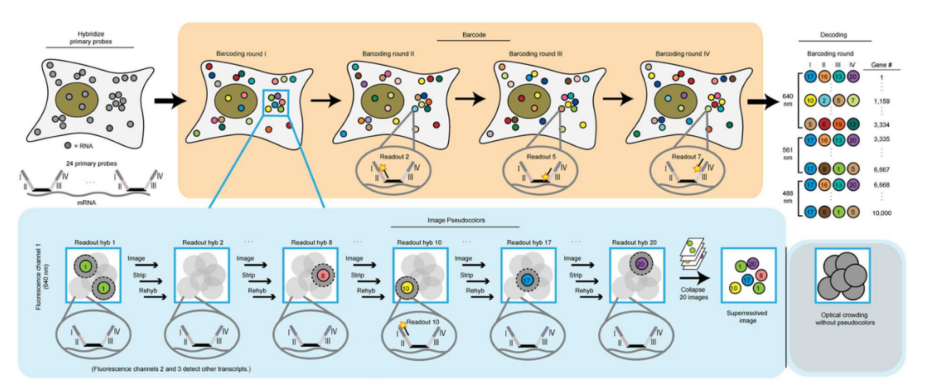
seqFISH+示意图
Split-FISH:生物组织中的高特异性多路RNA成像
Nature Methods [IF: 30.822]
① 提出了一种多重荧光原位杂交方法split-FISH,通过split探针的设计和优化,减少了杂交脱靶背景信号,大大降低了非透明化组织中的假阳性信号,实现更精确的RNA分析。
② 在A549细胞中使用粘蛋白5AC、寡聚粘液/凝胶形成(MUC5AC)转录本上的单分子FISH优化split探针序列,发现环形“桥”(bridge)具有更高的靶向信号且具有更高的特异性;优化split-FISH工作流程后,能够执行多次杂交和洗涤(至少20轮),而没有任何可观察到的FISH信号或RNA计数损失。
③ 通过量化单细胞中317个基因的分布和丰度,证明了split-FISH对各种小鼠组织(肾脏、肝脏、卵巢)的有效性,并揭示了复杂组织中转录组空间调节的不同定位模式。
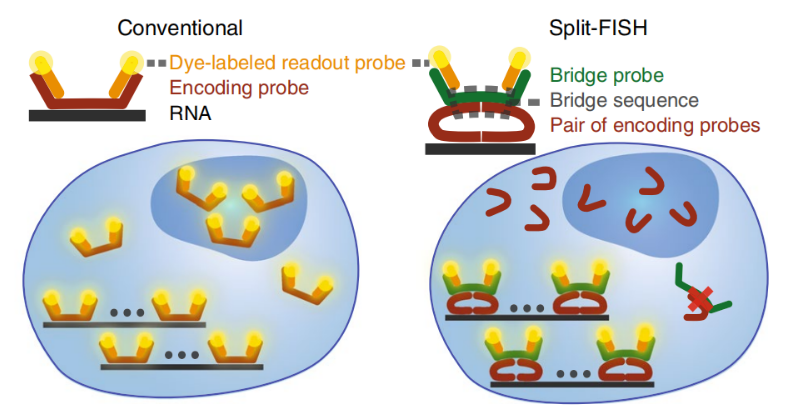
传统方法和split探针方法的比较
原位测序技术
In Situ Sequencing
STARmap:单细胞分辨率下3D完整组织原位RNA测序
Science [IF:41.058]
① 开发一种3D完整组织RNA测序技术——STARmap(spatially-resolved transcript amplicon readout mapping),它集成了水凝胶-组织化学、靶向信号放大和原位测序,为3D原位转录组学定义了一个平台。
② 将STARmap应用于小鼠大脑切片中,能以单细胞分辨率同时映射160~1,020个基因,证实该技术具有高效率、准确性和可重复性的功能特点。
③ 扩展到更大3D组织块,STARmap可识别和描述跨越立方毫米级(> 30,000个细胞)的兴奋性神经元亚型的分子定义的梯度分布,以及许多抑制性神经元亚型的短程3D自聚类。
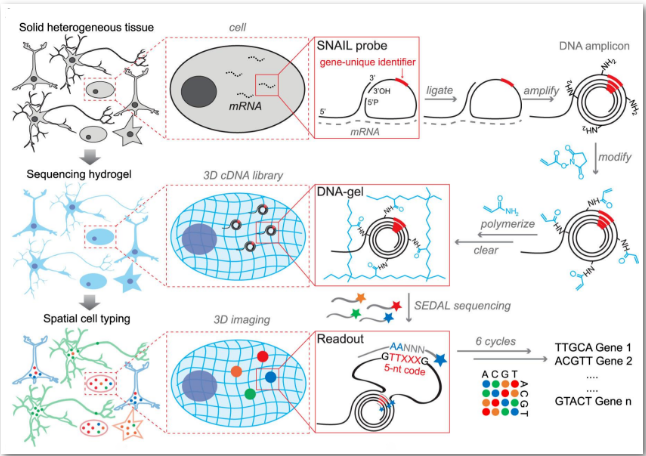
STARmap工作示意图
原位捕获技术
Next-Generation Sequencing with Spatial Barcoding
小鼠胚胎的单细胞空间转录组分析
Science [IF:49.962]
① 提出了一种将单细胞基因表达差异与空间背景结合起来的新方法——sci-Space,该方法能够在保留单细胞分辨率的同时在更大的尺度上解决空间异质性。
② 应用sci-Space对来自2个发育第14天的小鼠胚胎的14个矢状面切片进行了分析,绘制出121,909个细胞核的基因表达图谱和近似空间坐标,证明了sci-Space可以显示转录组中的任何基因,类似于原位杂交。
③ 通过分析图谱,鉴定出数千个基因呈现出与解剖结构有关的表达模式,在分析的细胞类型中结缔组织祖细胞和神经元的空间自相关基因最多,每个细胞中分别平均有12,150±2,270、8,623±3,846个基因,表明sci-Space能够区分由单个细胞类型驱动的基因表达空间模式,以及跨多个细胞类型的基因表达空间模式。
④ 利用空间信息对各种类型的细胞进行亚型注释,揭示了细胞类型在空间模式化程度方面存在很大差异,表明这些细胞亚型在体内以位置依赖的方式行事;采用拟时序分析,得出神经胶质细胞、辐射状细胞和神经元三种脑细胞随时间进行分化和迁移。
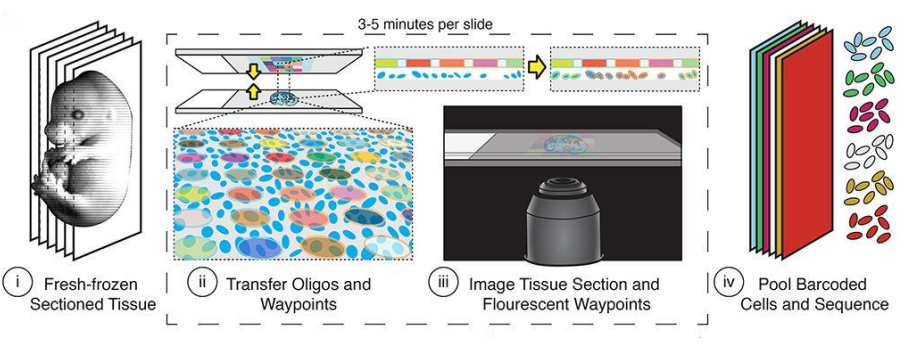
小鼠胚胎组织的sci-Space分析流程示意图
Seq-Scope:空间转录组显微检测
Cell [IF: 41.582]
① 基于空间条形码和测序平台,开发了可以实现亚微米分辨率的的空间转录组学新技术——Seq-scope,能较好地揭示基因空间表达信息,为空间单细胞和亚细胞分析提供了一个通用的解决方案。
② Seq-scope的中心到中心(center-to-center)分辨率为0.5~0.8 μm(平均约0.6 μm),可与光学显微镜相媲美,且具有高效的转录组捕获output(平均高达23~27 UMIs/μm2),当聚集到单细胞区域时,其转录组捕获output(平均4,700 UMIs/个细胞)基本与传统scRNA-seq相当。
③ 利用Seq-scope,清楚显示了肝脏和结肠的显微细胞和亚细胞结构的转录组图像,揭示了肝脏和结肠的空间转录组异质性。
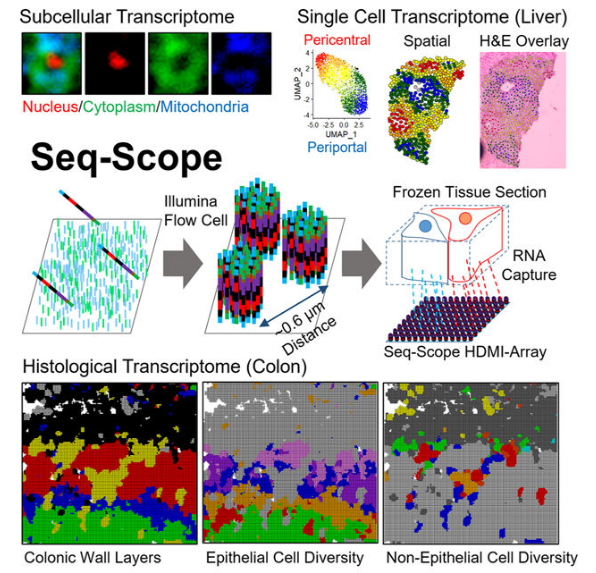
Seq-scope的工作流程图及应用
用于组织原位分析的高分辨空间转录组方法
Nature Method [IF: 28.547]
① 开发了高分辨率空间转录组学方法——HDST(High-Definition Spatial Transcriptomics),可以从含有密集空间条形码磁珠的组织切片中捕获RNA,用于空间分辨的基因表达分析,为研究健康和疾病组织结构和功能提供了更高分辨率的技术方法。
② HDST是将条形码的poly(d)T寡核苷酸沉积到2 μm孔中,通过顺序杂交和纠错策略解码它们的位置,然后将冷冻组织切片置于解码的载玻片上,染色和成像后捕获RNA,最后应用RNA-seq进行分析。
③ 将HDST应用于小鼠大脑的主嗅球(main olfactory bulb,MOB)和组织学三级乳腺癌HER2+患者的肿瘤切片中,证实了其是一种测量原位空间信息的高分辨方法,HDST的分辨率分别为ST(Spatial Transcriptomics)、Slide-Seq的1,400倍、25倍。
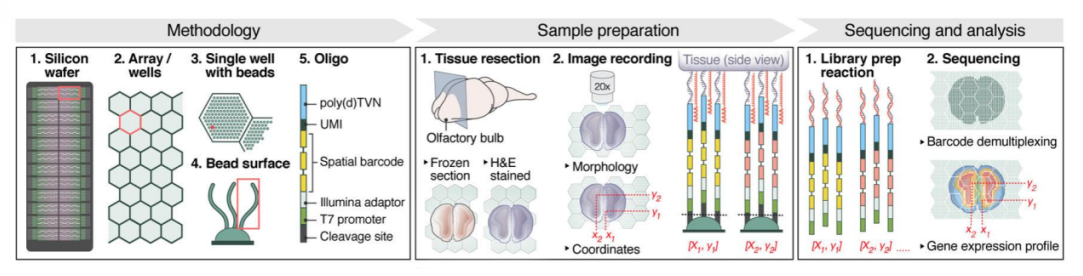
HDST工作流程示意图
关于时空组学技术Stereo-seq:
华大自主研发的时空组学技术Stereo-seq,是一项实现超高通量、超高精度的全景式时空转录组技术。Stereo-seq通过时空捕获芯片,结合原位RNA捕获,实现了500 nm的分辨率,最大捕获面积可达13 cm×13 cm,成为全球领先的能同时实现“纳米级分辨率”和“厘米级全景视场”的技术,且实现了基因与影像同时分析。与当前其他技术相比,在相同的精度下,Stereo-seq具备更灵敏和更强的mRNA捕获能力。
Stereo-seq目前已被成功应用到动物胚胎发育、植物发育、脑科学、肿瘤等多个不同领域。2022年5月,深圳华大生命科学研究院联合多家机构在Cell出版社官网以时空组学联盟(STOC)专题的形式发布了全球首批生命时空图谱,这是首次从时间和空间维度上对生命发育过程中的基因和细胞变化过程进行超高精度解析,为认知器官结构、生命发育、人类疾病和物种演化提供全新方向。除了生命时空图谱的绘制,时空组学技术Stereo-seq在疾病病理、脑科学等研究领域也发挥了重要作用。
版权声明:
本网站所有注明"来源:华大集团"或“来源:华大医疗”的文字,图片和音视频资料,版权均属于华大集团所有。非经授权,任何媒体,网站或个人不得转载,授权转载时须注明“来源:华大集团”。本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权,或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。




